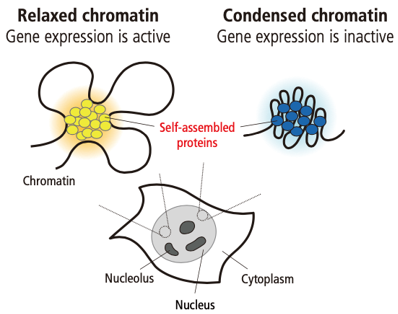
近年来,蛋白质或核酸通过自组装对细胞进行分隔并控制各种生物功能的 “相分离”现象已被报道并引起人们的关注。结构转换,如细胞核中染色质的解聚和浓缩,以及包括基因表达在内的核功能的控制机制,都开始被相分离所解释。本应用说明介绍了与日本癌症研究基金会癌症研究所的Ryu-Suke Nozawa博士合作进行的一个实验案例。他正试图通过分析作为各种核功能基础的染色质的结构以及控制染色质结构的分子机制来阐释多种生命现象的机制。
实验设备和软件:
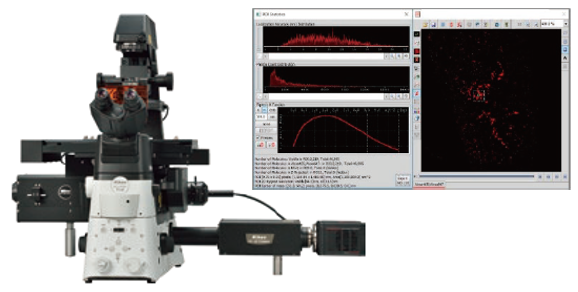
(1) 显微镜:Ti2-E (Nikon)
(2) 物镜:CFI SR HP Apochromat TIRF 100XC Oil
(3) 成像系统:N-STORM
(4) 图像软件:NIS-Elements (Nikon)
实验概要:
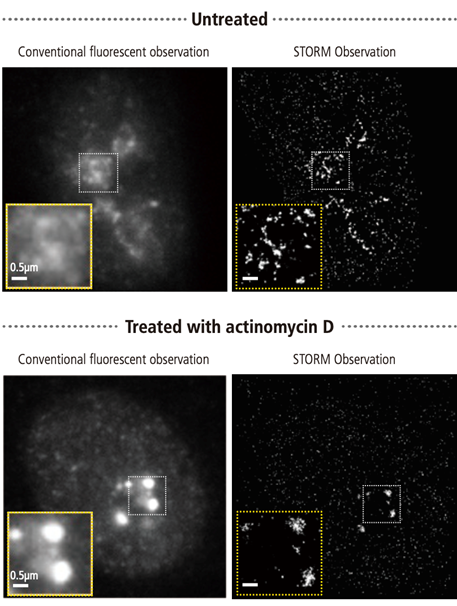
RNA聚合酶I是细胞核中核仁的主要组成蛋白之一,它从核糖体DNA转录核糖体RNA。在本实验中,通过使用尼康的超分辨率显微镜N-STORM,在单分子水平上观察RNA聚合酶I的定位(图2和图3)。然后,利用Actinomycin-D(放线菌素-D)处理以抑制RNA聚合酶I的功能,通过NIS-Elements的聚集分析功能对RNA聚合酶I分子的结构进行分析(图4)。
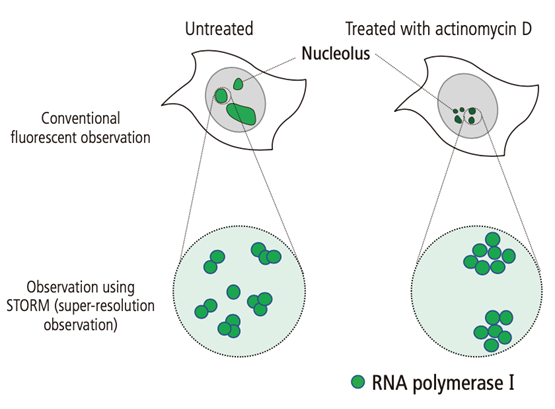
图2 定位于细胞核中的RNA聚合酶I。
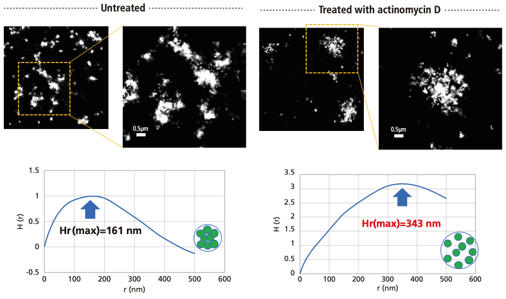
聚类分析的结果显示,当RNA聚合酶I的功能被Actinomycin-D处理所抑制时,与未处理的RNA聚合酶I相比,RNA聚合酶I的分子形成更大的聚合物(图4)。这表明,使用N-STORM的单分子成像与聚集分析作为一种新的“相分离”评价系统是切实可行的,因为它可以量化蛋白质的聚集和解聚程度。结果显示,RNA聚合酶I形成的簇的大小在未处理时为161nm,Actinomycin-D处理后为343nm。
N-STORM成像系统为研究带来的助益
1、使用N-STORM成像系统对样品进行观察,可获得传统光学显微镜获取不到的的超精细结构信息。
2、利用聚类分析功能,可实现在单分子水平上对目标蛋白的组装程度信息进行定量分析。
3、在单分子水平对目标蛋白进行聚集分析,是一种新的对“相分离”现象进行研究的有效手段。
4、从发现目标蛋白分子到获取STORM图像并进行分析,一系列的操作可被整合成为一个能够便利顺畅运行的工作流。
N-STORM成像系统及其聚集分析功能可作为研究“相分离”现象的新型评价系统,还可以在DNA-FISH实验中用于染色质的可视化及结构分析。此外,得益于其多色成像的能力,N-STORM还可用于分析干细胞分化过程中的染色质变化、多种细胞调控因子间的相关性分析。
产品信息:
(1) N-STORM超分辨率显微镜:N-STORM利用一种名为STochastic Optical Reconstruction Microscopy(STORM)的定位技术,实现了约为传统光学显微镜十倍的分辨率。它允许在分子水平上对细胞器的结构进行成像。其横向分辨率约为20纳米,轴向分辨率约为50纳米。
(2) 聚类分析 (Cluster Analysis) 功能:通过在超分辨率显微镜捕获的图像中用ROI指定目标区域来自动分析荧光点的分布模式。随着定量评估加入到纳米级精度的图像采集中,从图像中获得的信息的可靠性得到了显著提高。

– The End –

