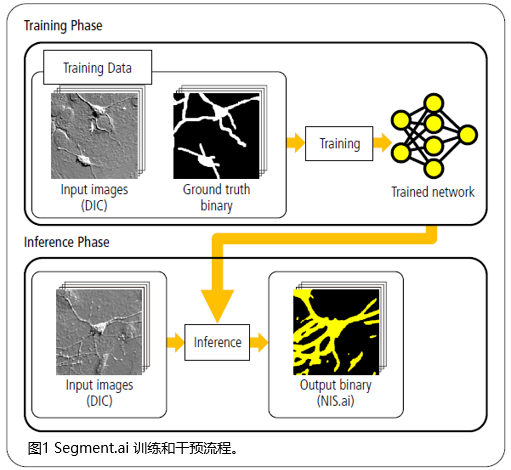
当共同培养多种类型的细胞时,一般需要分子标签进行细胞标记,以识别特定的细胞类型 [1] 。然而,免疫染色需要固定细胞,这与活体观察是不相容的。此外,在使用荧光蛋白进行活体细胞观察时,转基因和荧光标记产生的毒副作用可能会成为一个问题。 NIS.ai ( Segment.ai) 是 NIS-Elements成像软件的一个深度学习模块,是一项用于检测传统的二值化和图像处理难以提取的特定细胞区域的技术。这种方法使从未标记的图像中检测细胞类型成为可能。
本应用手册展示了如何使用 Segment.ai模块识别和定量分析微分干涉图像( DIC)中共同培养的神经元和星形胶质细胞区域。该研究是由国立自然科学研究院生命系统探索研究中心主任 Tomomi Nemoto博士和生物光子学研究小组的 Hirokazu博士与 Motosuke Tsutsumi博士合作完成。 实验方法
共同培养 10天的神经元和星形胶质细胞用 4%多聚甲醛固定,然后用突触蛋白 (synaptophysin,神经元 marker) 抗体进行免疫染色。采集样品 DIC图像作为训练和推断的图像依据。从获得的 DIC图像中,创建了一个总细胞区域的真值二进制图像(注释)和一个根据荧光标记 synaptophysin 推断的神经元区域,用来做训练用数据( 40套)。 使用这些数据,利用 Segment.ai 模块进行了 1000 次迭代训练,以创建一个基于 DIC 图像细胞区域检测的 Segment.ai 模型(图 1 )。
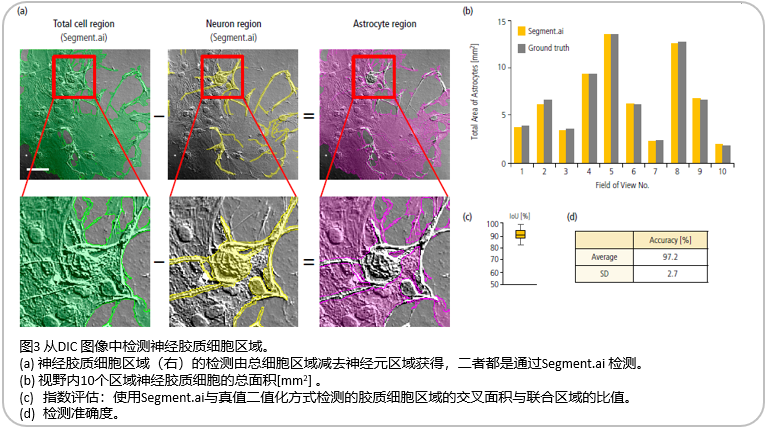
通过应用上述经过训练的 Segment.ai模型,我们在共同培养神经元和星形胶质细胞的 DIC 图像中检测不同细胞的区域(图 2a)。 通过检测神经元的长度,对 Segment.ai 检测到的神经元区域和手动选取的真值神经元区域的准确性进行了比较,发现其准确率为 82.4% (图 2b, )。 此外,我们还尝试了检测星形胶质细胞区域,方法是使用 Segment.ai 将神经元区域从总细胞区域中减去(图 3a )。结果显示,相对于真值数据,使用 Segment.ai 方式星形胶质细胞区域也以较高的精度被量化( 97.2% )(图 3b, 3c, 3d )。
讨论和总结
通过使用 NIS.ai,每个区域的神经元和星形胶质细胞都可以通过输入 DIC图像来识别, 可高精度量化神经元的长度和细胞面积。 有关神经元的研究可能需要超过 10天的长期培养 [2], 由于 Segment.ai方法对神经元的延时观 察进行无标记的分析,因此可最小化对培养实验的影响,避免了标记引起的细胞毒性。 参考文献
1. N. Goshi et a/., “A primary neural cell culture model to study neuron, astrocyte, and microglia interactions in neuroinflammation” . J. Neuroinflammation. 17:155. (2020). 2. R.T. Roppongi et a/., “Low-density primary hippocampal neuron culture”. JoVe . doi:10.3791/55000. (2017).
产品信息
NIS.ai 显微成像模块
NIS.ai 图像处理 /分析模块,扩展了 NIS-Elements成像软件的功能,提供四种 AI模块类型: Enhance.ai, Convert.ai, Segment.ai 和 Clarify.ai。 本应用手册中使用的 Segment.ai 模块可以对您需要分析的细胞和结构进行分割提取。该 AI 模块为传统的二进制化难以提取的目标和以前需要手动分类的目标提供了新的解决方案。
– The End –
收藏
文章导航